| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 |
| 8 | 9 | 10 | 11 | 12 | 13 | 14 |
| 15 | 16 | 17 | 18 | 19 | 20 | 21 |
| 22 | 23 | 24 | 25 | 26 | 27 | 28 |
- RNN
- 파이썬
- 시그모이드
- 이항분포
- 결정트리
- 블록체인
- 인공지능 수학
- 바이오인포매틱스
- Java
- SVM
- HMM
- 생물정보학
- 인공신경망
- 딥러닝
- 오류역전파
- CNN
- MERS
- 서열정렬
- Kaggle
- 자바
- bioinformatics
- 생명정보학
- 인공지능
- AP
- 바이오파이썬
- COVID
- BLaST
- AP Computer Science A
- 캐글
- ncbi
- Today
- Total
데이터 과학
Jmol 설치 본문
GUI 기반 분자 모델링 프로그램 Jmol 설치입니다.
Jmol: an open-source Java viewer for chemical structures in 3D
Those with interest in molecular visualization, especially the education and research communities, are encouraged to join the jmol-users mailing list or even the jmol-developers mailing list. There you can share ideas and experiences, ask for help, give us
jmol.sourceforge.net
jmol 사이트 접속하여서 다운로드합니다.
소스포지 사이트입니다.

최신버전을 다운로드 받습니다.
16.1.41이며 압축파일로 되어 있는데 압축을 풀면 다음과 같은 목록이 나옵니다.

여기서 .bat 파일인 jmol.bat 파일 (windows 배치 파일)을 실행하면 됩니다.
만약 실행이 안되면 JAVA를 설치(https://www.oracle.com/java/technologies/)해야 합니다.
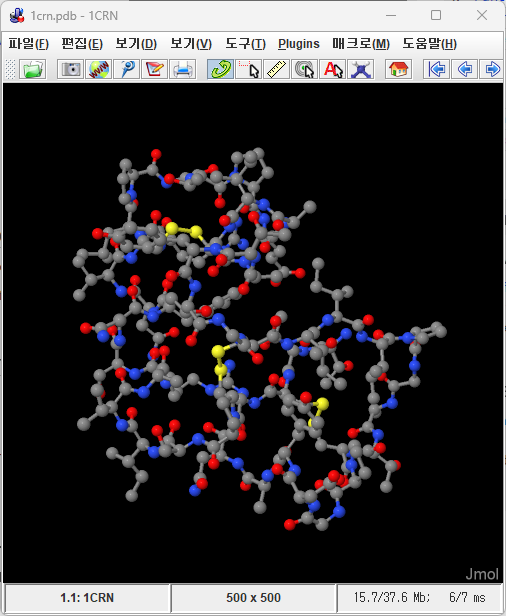
배치 파일을 실행하면 다음과 같은 화면이 나타나는데 Jmol 프로그램입니다.

저는 자바 버전이 1.8이네요.
프로그램에서 파일-> Get PDB를 선택하면 1 crn이 기본으로 설정되어 있습니다.
버튼을 누르면 다음과 같은 화면이 나탑니다.
https://www.ebi.ac.uk/pdbe/entry/pdb/1crn/index
PDB 1crn structure summary ‹ Protein Data Bank in Europe (PDBe) ‹ EMBL-EBI
The sliders below show the change in model quality between original PDB entry and the PDB-REDO entry
www.ebi.ac.uk
'생명정보학 & 화학정보학 > MEGA and Chimera' 카테고리의 다른 글
| scratch, 프로테오믹스 (0) | 2023.09.16 |
|---|---|
| Hmmer 사용법 - 리눅스 (0) | 2022.11.22 |
| ucsf 키메라(chimera) 사용법 (0) | 2022.11.15 |
| WEKA 사용법 (0) | 2021.10.09 |
| MEGA-X 사용법 (0) | 2021.05.23 |



