| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 |
| 8 | 9 | 10 | 11 | 12 | 13 | 14 |
| 15 | 16 | 17 | 18 | 19 | 20 | 21 |
| 22 | 23 | 24 | 25 | 26 | 27 | 28 |
- CNN
- 딥러닝
- 오류역전파
- 바이오파이썬
- 캐글
- 결정트리
- bioinformatics
- AP Computer Science A
- BLaST
- HMM
- MERS
- RNN
- ncbi
- Java
- 자바
- 바이오인포매틱스
- 인공신경망
- 블록체인
- COVID
- AP
- 이항분포
- 인공지능 수학
- 파이썬
- 생물정보학
- 인공지능
- 서열정렬
- 생명정보학
- Kaggle
- SVM
- 시그모이드
- Today
- Total
데이터 과학
MEGA-X 사용법 본문
MEGA-X를 다운 받는다. (https://www.megasoftware.net/)
File 메뉴에서 open a File/seesion ... 메뉴를 선택한다.
저장했던 FASTA 형식으로 된 파일을 불러온다.
FASTA 파일(https://tsyoon.tistory.com/2)은 이전 BIOEDIT에서 저장했던 파일을 사용해도 된다.
Analyze or Align File? 이라는 팝업 메뉴가 뜨는데 여기서 Align을 선택한다.

Analyze를 선택하게 되면 Editor 메뉴가 뜨는데 형식을 변경하고자 할 때 사용 할 수 있다.

Align을 선택해서 서열 정리된 화면을 한번 보도록 하자.
Aligment에서 ClustalW, MUSCLE 메뉴들을 선택할 수 있다.
ClustalW를 선택한 후 정렬하면


서열정렬이 나타나는데 앞서 bioEdit에서 진행했던 서열과 큰 차이는 없다. ClustalW 알고리즘이 같다.
계통도 분석을 위한 방법은 다음과 같다.
Data -> Phylogeny Analysis 메뉴를 선택한다.
*.meg 파일로 저장을 하고 MEGA 화면에서 Phylogeny -> Constructor 메뉴를 선택한다.

OK를 선택하면 다음과 같은 결과가 나타난다.
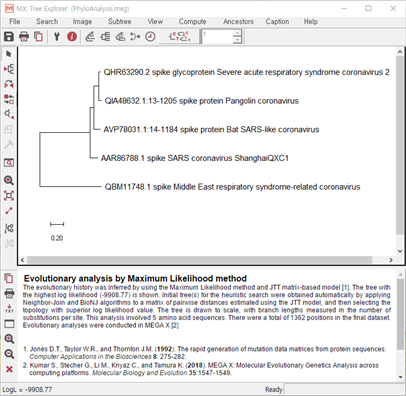
이 계통도를 통해 현재 SARS2와 Pangolin의 관계를 알 수 있으며, MERS와는 거리가 있다는 것을 분석 할 수 있다.
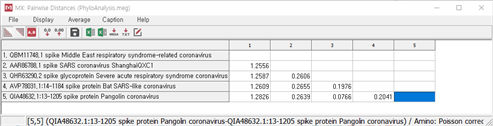
Distance -> Compute Pairwise Distances...를 선택하면 거리값을 나타낼 수 있다,
서열정렬 후에 MEGA format으로 저장하는 방법은 다음과 같다.
Data -> Export Alignment -> Mega format
알맞은 파일이름으로 입력 한 후 저장한다.
다양한 계통수 알고리즘으로 작성이 가능하다.

계통수 (Tree) 메뉴는 5가지의 메뉴가 있다.
Maximun likelihood Tree
Neighbor-Joining Tree
Minimum-Evolution Tree
UPGMA(Unweighted Pair Group Method 및 Arithmetic Mean) Tree
Maximum Parsimony Tree
BLAST
BLAST 사이트에 접속한 후 https://blast.ncbi.nlm.nih.gov/Blast.cgi
하단에 있는 Multiple Alignment를 선택한다.
MEGA-X에서 사용했던 FASTA 서열을 복사한 후 입력한다. Align을 클릭한다.
결과는 Alignment, Description, Graphical Overview까지 나타난다.
블라스트 사용법 참고 ->
위에 있는 메뉴중에 Phylogenetic Tree를 선택하면 계통수 나무가 결과로 보인다.

Mega-x에서 나온 결과와 큰 차이점은 없다.
download를 선택하면 Fasta plus gaps, Clustal, Phylip, Nexux, ASN.1 파일 형식으로 다운로드 받을 수 있다.
'생명정보학 & 화학정보학 > MEGA and Chimera' 카테고리의 다른 글
| scratch, 프로테오믹스 (0) | 2023.09.16 |
|---|---|
| Hmmer 사용법 - 리눅스 (0) | 2022.11.22 |
| ucsf 키메라(chimera) 사용법 (0) | 2022.11.15 |
| WEKA 사용법 (0) | 2021.10.09 |
| BIO EDIT 사용법 (0) | 2021.05.22 |



