| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 |
| 8 | 9 | 10 | 11 | 12 | 13 | 14 |
| 15 | 16 | 17 | 18 | 19 | 20 | 21 |
| 22 | 23 | 24 | 25 | 26 | 27 | 28 |
- CNN
- 딥러닝
- 결정트리
- ncbi
- bioinformatics
- 캐글
- 인공지능 수학
- 바이오파이썬
- 블록체인
- 서열정렬
- Kaggle
- AP
- 이항분포
- 오류역전파
- 시그모이드
- Java
- 생명정보학
- 생물정보학
- 파이썬
- 바이오인포매틱스
- RNN
- MERS
- BLaST
- 자바
- SVM
- COVID
- 인공신경망
- 인공지능
- AP Computer Science A
- HMM
- Today
- Total
데이터 과학
2021버전 SARS-CoV-2 핵산서열 분석 본문
Kaggle에서 예제로 사용되는 MN908947.fasta는 2020년 코로나 바이러스 버전으로 변이가 일어난 2021년 데이터와는 다릅니다. 그래서 현재 변이를 주도하는 코로나 핵산서열을 가지고 간단히 실험을 진행해 보겠습니다.
https://www.ncbi.nlm.nih.gov/nuccore/MZ021506.1
Severe acute respiratory syndrome coronavirus 2 isolate SARS-CoV-2/hum - Nucleotide - NCBI
no features Feature First Previous Next Last Details
www.ncbi.nlm.nih.gov
위 파일을 다운로드 받아 주피터 노트북에 업로드 해 봅시다.
이제는 SeqRecord 함수를 이용해서 데이터가 잘 올라가 있는지 확인합니다.
>>>from Bio import SeqIO
>>>for sequence in SeqIO.parse('MZ021506.1.fasta', "fasta"):
>>> print('Id: ' + sequence.id + '\nSize: ' + str(len(sequence))+' nucleotides')
Id: MZ021506.1
Size: 29805 nucleotides
>>>DNAseq = SeqIO.read('MZ021506.1.fasta', "fasta")
>>>DNA = DNAseq.seq
>>>mRNA = DNA.transcribe()
>>>Amino_Acid = mRNA.translate()
>>>Proteins = Amino_Acid.split('*')
>>>Proteins
[Seq('NKPTNFRSLVDLFSKRTLKSVWLSLGCMLSALTQYN'), Seq(''), Seq('LITVVDRTRVTRLSSAGCLRFRPFCSRSSAHLGFVRV'), Seq('PKGKMESLVPGFNEKTHVQLSLPVLQVRDVLVRGFGDSVEEVLSEARQHLKDGT...FAV'), Seq('VQPVLHRAAQALVLMSYTGLLTSTMIK'), Seq('LVLLNS'),
....
이제 아미노산 서열에서 *로 표시된 정지코돈으로 나타낸 것을 지우면서 서열의 20개 미만인 아미노산은 제거해 봅니다.
>>>for i in Proteins[:]:
>>> if len(i) < 20:
>>> Proteins.remove(i)
>>>Proteins
[Seq('NKPTNFRSLVDLFSKRTLKSVWLSLGCMLSALTQYN'), Seq('LITVVDRTRVTRLSSAGCLRFRPFCSRSSAHLGFVRV'), Seq('PKGKMESLVPGFNEKTHVQLSLPVLQVRDVLVRGFGDSVEEVLSEARQHLKDGT...FAV'), Seq('VQPVLHRAAQALVLMSYTGLLTSTMIK'), Seq('LRDTLSLTTNMKKQFIIYLRIVQLLLNMTSLSLE'), Seq('TVTWYHIYHVNVLLNTQWQTSSML'), Seq('KKYLSHTIVVMMIISIKRTGMIL'),
....
이제 Pandas 라이브러리와 ProtPatam을 이용해서 Moleclular Weights 값을 구해 봅시다.
>>>import pandas as pd
>>>from Bio.SeqUtils.ProtParam import ProteinAnalysis
>>>MW = []
>>>aromaticity =[]
>>>AA_Freq = []
>>>IsoElectric = []
>>>for j in Proteins[:]:
>>> a = ProteinAnalysis(str(j))
>>> MW.append(a.molecular_weight())
>>> aromaticity.append(a.aromaticity())
>>> AA_Freq.append(a.count_amino_acids())
>>> IsoElectric.append(a.isoelectric_point())
>>>MW = pd.DataFrame(data = MW,columns = ["Molecular Weights"] )
>>>MW.head()
Molecular Weights
| 4132.8054 |
| 4164.8639 |
| 490392.0277 |
| 2942.5413 |
| 4010.8050 |
위치적으로 보면 3번째 위치에 있는 MW 값이 상당히 높아보입니다.
pyplot과 seaborn으로 그래픽화 해서 결과를 확인해 보겠습니다.
>>>import matplotlib.pyplot as plt
>>>import seaborn as sns
>>>sns.set_style('whitegrid');
>>>plt.figure(figsize=(10,6));
>>>sns.distplot(MW,kde=False);
>>>plt.title("SARS-CoV-2 Protein Molecular Weights Distribution");
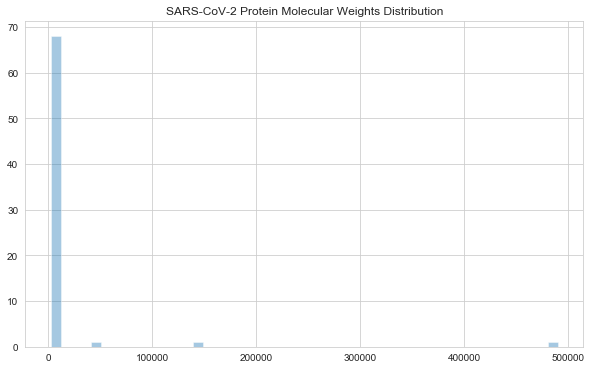
MW 값을 막대그래프로 나타낸 결과를 보니 500000에 가까운 값이 하나 있는 것을 확인 할 수 있네요.
>>>MW.idxmax()
Molecular Weights 2
dtype: int64
가장 큰 값을 가진 데이터 위치는 세번째입니다. [2]
>>>print(Proteins[2])
>>>len(Proteins[2])
PKGKMESLVPGFNEKTHVQLSLPVLQVRDVLVRGFGDSVEEVLSEARQHLKDGTCGLVEVEKGVLPQLEQPYVFIKRSDARTAPHGHVMVELVAELEGIQYGRSGETLGVLVPHVGEIPVAYRKVLLRKNGNKGAGGHSYGADLKSFDLGDELGTDPYEDFQENWNTKHSSGVTRELMRELNGGAYTRYVDNNFCGPDGYPLECIKDLLARAGKASCTLSEQLDFIDTKRGVYCCRE......
4409
위치는 [2]이기에 3번째에 있는 서열이고 그 길이는 4409개입니다.
이전에 실험했던 이전 데이터와는 그 길이 차이가 크네요. 이전 데이터인 MN908947.fna에서는 2701개입니다. 약 2배 정도 서열이 늘어나 있다는 것을 확인 할 수 있습니다.
https://tsyoon.tistory.com/17 (MN908947.fna 실험)
바이오파이썬 단백질 분석 : COVID-19 게놈(Genome)
이번 내용은 바이오파이썬으로 단백질 분석하는 내용입니다. 파이썬 도크(https://github.com/kaggle/docker-python)에 대한 소개를 하면서 필요할 때는 numpy와 pandas 라이브러리를 이용해서 분석하는 방법
tsyoon.tistory.com
4409개와 2701개의 염기서열 갯수는 큰 차이입니다. 전염력과 관계가 있을까요?
aromaticity와 isoelectric point에 대해서도 값이 얼마인지 알아 봅시다.
>>>print('The aromaticity % is ',aromaticity[2])
>>>print('The Isoelectric point is', IsoElectric[2])
The aromaticity % is 0.10183715128146972
The Isoelectric point is 6.0700292587280265
새로운 핵산 서열이 나오면 비교해 보면사 이전 서열들과 어떤 변화가 있는지 확인 할 수 있습니다. 최대 서열 길이가 많이 바뀌어 있습니다. 어떤 의미가 있을까요?
'생명정보학 & 화학정보학 > 바이오파이썬' 카테고리의 다른 글
| 블라스트 XML에서의 검색 (0) | 2021.08.29 |
|---|---|
| 바이오파이썬에서 계통수 그리기 (0) | 2021.08.22 |
| GenBank 정보 불러오기 (2) | 2021.08.15 |
| 바이오파이썬 단백질 분석 : COVID-19 게놈(Genome) (1) | 2021.07.27 |
| COVID-19: Biopython으로 단백질 식별하는 방법 (Kaggle) (1) | 2021.07.18 |


